5-メチルシトシン
4-amino-5-methyl-3H-pyrimidin-2-one
識別情報
CAS登録番号 554-01-8
PubChem
65040
ChemSpider
58551
UNII
6R795CQT4H
KEGG
C02376
MeSH
5-Methylcytosine
ChEBI
O=C1/N=C\C(=C(\N)N1)C
Cc1cnc(=O)[nH]c1N
InChI=1S/C5H7N3O/c1-3-2-7-5(9)8-4(3)6/h2H,1H3,(H3,6,7,8,9)
Key: LRSASMSXMSNRBT-UHFFFAOYSA-N
InChI=1/C5H7N3O/c1-3-2-7-5(9)8-4(3)6/h2H,1H3,(H3,6,7,8,9)
Key: LRSASMSXMSNRBT-UHFFFAOYAO
特性
化学式
C5 H7 N3 O
モル質量
125.13 g mol−1
特記なき場合、データは常温 (25 °C )・常圧 (100 kPa) におけるものである。
5-メチルシトシン (5mC、m5Cなどと略されることが多い)またはC5-メチルシトシン とは、DNA 塩基 の一つであるシトシン(C) がメチル化 されたものである。
遺伝子 の転写 調整を始めとする様々な生命現象等に関与している[ 1] 遺伝子発現 に変化が生ずる例が知られており、この分野の研究はエピジェネティクス と呼ばれる。
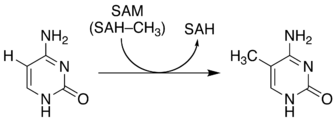
5mCはDNAメチルトランスフェラーゼ によるエピジェネティック な修飾により生成される。5mCとヌクレオシド が組み合わさった状態は5-メチルシチジン であり、この状態でDNAに組み込まれている。
5mCではメチル基 は六員環 の、図の6時方向の窒素 原子(NH)から反時計回りに数えて5位の炭素原子に付加される。このメチル基は、シトシンと5mCとを区別する特徴である。
1898年、結核菌 から細菌 性毒素 を単離しようとしていた時、新たな核酸が発見され、ツベルクリン酸 (英語版 ) [ 2] チミン 、グアニン 、シトシン とは異なる、メチル化された塩基を持つヌクレオチド であった。1925年、少量のメチル化シトシンがツベルクリン酸の硫酸加水分解 物として生成された[ 3] [ 4] [ 5] 胸腺 のDNA から従来のシトシンやウラシル とは異なるメチル化シトシンがペーパークロマトグラフィー により単離され、存在が決定的となった[ 6]
それから70年後、RNA 分子中に一般に存在することが明らかとなったが、正確な役割は不明であった[ 7]
In vivo この物質の機能は生物種により大きく異なる[ 8]
細菌の場合、5mCはゲノム上の様々な場所に存在し、しばしば自身のDNAをメチル化感受性制限酵素 から保護するマーカーとして機能している。
植物の場合、5mCはCpGサイト 、CpHpG、ならびにCpHpHの配列に局在している(H = A, C, T)。
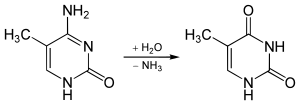
真菌および動物の場合、5mCは主にCpGジヌクレオチドとして存在する。多くの真核生物 ではCpGサイトのメチル化率はわずかであるが、脊椎動物 の場合は70〜80%のCpG中シトシンがメチル化されている[ 9] [ 10] シトシン (C)が自発性に脱アミノ化 するとウラシル (U)となり、DNA修復酵素 により除去されるが、一方で5mCが脱アミノ化するとチミン (T)を生じる。この塩基の転換(C→T)は塩基転位型突然変異 を引き起こし得る[ 11] APOBEC (英語版 ) [ 12] 5-ヒドロキシメチルシトシン の脱アミノ化の意義や詳細は不明である。
In vitro 亜硝酸 等により5-メチルシトシンから-NH2 基を除去(脱アミノ化)するとチミン を生ずる。同じ条件下でシトシンが脱アミノ化するとウラシル (U)を生じる。
5-メチルシトシンからの脱アミノ化に拠るチミンの生成 シトシンは重亜硫酸 処理で脱アミノ化されるが、5mCは脱アミノ化されない。この性質を利用して、重亜硫酸塩シークエンス (英語版 ) [ 13]
5mCのメチル基は、DNAメチルトランスフェラーゼ (DNMT)を介してゲノムDNA中のシトシンに付加される。ヒトゲノム中では、DNMT1 、DNMT2、DNMT3A 、DNMT3B 、およびDNMT3L の5つのDNMTが知られている。藻類および菌類には、DNMT4、DNMT5、およびDNMT6の3つがさらに存在する[ 14]
DNMT1には、RFTS(replication foci targeting sequence)と、5mCマークの追加を触媒するCXXCドメインが含まれている。RFTSはDNMT1をDNA複製の遺伝子座に導き、DNA複製中の娘鎖上の5mCの維持を支援する。CXXCはDNAへのメチル化のde novo 付加のためのジンクフィンガー ドメインを含む [ 15] [ 16] de novo [ 17] [ 18] [ 16]
Addition of methyl group to cytosine 付加のメカニズムは次のとおりである [ 19]
DNMTのPCQモチーフのシステイン残基が、メチル化されるシトシンヌクレオチドの炭素6に求核攻撃を引き起こす。
S-アデノシルメチオニン は、メチル基を炭素5に供与する。DNMT酵素の塩基は、5位の炭素の残留水素を脱プロトン化して、5位と6位の炭素間の二重結合を復元し、5-メチルシトシン塩基対を生成する。
シトシンが5mCにメチル化された後、複数のメカニズムを介して元の状態に戻すことができる。
受動的なDNA脱メチル化としては、DNMTによるメンテナンスを抑制(あるいは欠如)することで、ゲノム複製を通じて徐々にメチル化シトシンを希釈化する、というものがある。能動的なDNA脱メチル化としては、酸化プロセスにより5-ヒドロキシメチルシトシン (5hmC)、5-ホルミルシトシン (5fC)、および5-カルボキシルシトシン (5caC)に変換され、後者2つは最終的にチミンDNAグリコシラーゼ (TDG)及びシトシンを復元するための塩基除去修復酵素(BER)によってヌクレオシドから切断される[ 20] [ 21]
5mC、5hmC、および5fCへの酸化反応はTET (Ten-eleven translocation)ファミリージオキシゲナーゼを介して発生する。この酵素は5mCを優先的に処理し、TET2では5hmCおよび5fC変換の初期反応速度は4.9〜7.6倍遅くなる[ 22] α-ケトグルタル酸 (α-KG)が必要であり、後者の基質はイソクエン酸デヒドロゲナーゼ (IDH)によってイソクエン酸 から生成される[ 23] 悪性腫瘍 (癌細胞)においては、α-KGと競合する2-ヒドロキシグルタレート (2HG)を生成してTET活性を低下させることで、5mCから5hmCへの変換を低下させている可能性が報告されている[ 24]
癌細胞においては、ゲノムDNAは過剰メチル化状態や低メチル化状態になることがある[ 25] de novo メチル化されることで、通常腫瘍の増殖阻害に関連する遺伝子の異常な不活性化を引き起こすものがある[ 26] [ 27]
サテライトDNA 、Alu配列、および長い散在反復配列(LINE)を含むゲノム中の反復配列は、癌細胞において低メチル化されていることが多く、通常は発現しないはずの遺伝子の発現をもたらすため、その発現レベルは多くの場合で腫瘍進行の重要なマーカーになっている[ 25]
低メチル化領域における遺伝子の過剰発現は、ゲノム全体の高メチル化により抑制される可能性もある[ 25] [ 28]
「Epigenetic age」とは、年齢とDNAメチル化レベルとの関係を指す[ 29]
エピジェネティックな年齢の推定方法は複数提唱されている。「Horvathの時計」では、複数組織由来の353個のCpGセットを測定する。このセットでは、半分は年齢と正の相関があり、残りの半分はエピジェネティックな年齢を推定するために負の相関がある[ 30] [ 31] [ 32] [ 33]
^ Wu, Xiaoji; Zhang, Yi (09 2017). “TET-mediated active DNA demethylation: mechanism, function and beyond” . Nature Reviews. Genetics 18 (9): 517–534. doi :10.1038/nrg.2017.33 . ISSN 1471-0064 . PMID 28555658 . https://www.ncbi.nlm.nih.gov/pubmed/28555658 . ^ Matthews AP (2012). Physiological Chemistry RareBooksClub.com . pp. 167. ISBN 1130145379 ^ Johnson TB, Coghill RD (1925). “The discovery of 5-methyl-cytosine in tuberculinic acid, the nucleic acid of the Tubercle bacillus ”. J Am Chem Soc 47 (11): 2838–2844. doi :10.1021/ja01688a030 . ^ Grosjean H (2009). Nucleic Acids Are Not Boring Long Polymers of Only Four Types of Nucleotides: A Guided Tour . Landes Bioscience.
^ Vischer E, Zamenhof S, Chargaff E (1949). “Microbial nucleic acids: the desoxypentose nucleic acids of avian tubercle bacilli and yeast”. J Biol Chem 77 (1): 429–438. PMID 18107446 . ^ Hotchkiss RD (1948). “The quantitative separation of purines, pyrimidines and nucleosides by paper chromatography”. J Biol Chem 175 (1): 315–332. PMID 18107446 . ^ Squires JE, Patel HR, Nousch M, Sibbritt T, Humphreys DT, Parker BJ, Suter CM, Preiss T. (2012). “Widespread occurrence of 5-methylcytosine in human coding and non-coding RNA ”. Nucleic Acids Res 40 (11): 5023–5033. doi :10.1093/nar/gks144 . PMID 22344696 . ^ Colot V, Rossignol JL (1999). “Eukaryotic DNA methylation as an evolutionary device”. Bioessays 21 (5): 402–411. doi :10.1002/(SICI)1521-1878(199905)21:5<402::AID-BIES7>3.0.CO;2-B . PMID 10376011 . ^ Bird, A. P. (1986 May 15-21). “CpG-rich islands and the function of DNA methylation” . Nature 321 (6067): 209–213. doi :10.1038/321209a0 . ISSN 0028-0836 . PMID 2423876 . https://www.ncbi.nlm.nih.gov/pubmed/2423876 . ^ Ehrlich, M.; Wang, R. Y. (1981-06-19). “5-Methylcytosine in eukaryotic DNA” . Science (New York, N.Y.) 212 (4501): 1350–1357. doi :10.1126/science.6262918 . ISSN 0036-8075 . PMID 6262918 . https://www.ncbi.nlm.nih.gov/pubmed/6262918 . ^ Sassa, A; Kanemaru, Y; Kamoshita, N; Honma, M; Yasui, M (2016-09-01). “Mutagenic consequences of cytosine alterations site-specifically embedded in the human genome” (英語). Genes and Environment 38 (1). doi :10.1186/s41021-016-0045-9 . ISSN 1880-7062 . PMC 5007816 . PMID 27588157 . https://genesenvironment.biomedcentral.com/articles/10.1186/s41021-016-0045-9 . ^ Chahwan R., Wontakal S.N., and Roa S. (2010). “Crosstalk between genetic and epigenetic information through cytosine deamination”. Trends in Genetics 26 (10): 443–448. doi :10.1016/j.tig.2010.07.005 . PMID 20800313 . ^ Clark SJ, Harrison J, Paul CL, Frommer M (1994). “High sensitivity mapping of methylated cytosines” . Nucleic Acids Res. 22 (15): 2990–2997. doi :10.1093/nar/22.15.2990 . PMC 310266 . PMID 8065911 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC310266/ . ^ Ponger, Loïc; Li, Wen-Hsiung (2005-04-01). “Evolutionary Diversification of DNA Methyltransferases in Eukaryotic Genomes” (英語). Molecular Biology and Evolution 22 (4): 1119–1128. doi :10.1093/molbev/msi098 . ISSN 0737-4038 . PMID 15689527 . https://academic.oup.com/mbe/article/22/4/1119/1083517 . ^ Lyko, Frank (February 2018). “The DNA methyltransferase family: a versatile toolkit for epigenetic regulation” (英語). Nature Reviews Genetics 19 (2): 81–92. doi :10.1038/nrg.2017.80 . ISSN 1471-0064 . PMID 29033456 . ^ a b Robertson, K D; Uzvolgyi, E; Liang, G; Talmadge, C; Sumegi, J; Gonzales, F A; Jones, P A (1999-06-01). “The human DNA methyltransferases (DNMTs) 1, 3a and 3b: coordinate mRNA expression in normal tissues and overexpression in tumors.” . Nucleic Acids Research 27 (11): 2291–2298. doi :10.1093/nar/27.11.2291 . ISSN 0305-1048 . PMC 148793 . PMID 10325416 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC148793/ .
^ Wu, Xiaoji; Zhang, Yi (2017-05-30). “TET-mediated active DNA demethylation: mechanism, function and beyond”. Nature Reviews Genetics 18 (9): 517–534. doi :10.1038/nrg.2017.33 . ISSN 1471-0056 . PMID 28555658 . ^ Jia, Da; Jurkowska, Renata Z.; Zhang, Xing; Jeltsch, Albert; Cheng, Xiaodong (September 2007). “Structure of Dnmt3a bound to Dnmt3L suggests a model for de novo DNA methylation” (英語). Nature 449 (7159): 248–251. Bibcode : 2007Natur.449..248J . doi :10.1038/nature06146 . ISSN 1476-4687 . PMC 2712830 . PMID 17713477 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC2712830/ . ^ Lyko, Frank (February 2018). “The DNA methyltransferase family: a versatile toolkit for epigenetic regulation” (英語). Nature Reviews Genetics 19 (2): 81–92. doi :10.1038/nrg.2017.80 . ISSN 1471-0064 . PMID 29033456 . ^ Wu, Xiaoji; Zhang, Yi (2017-05-30). “TET-mediated active DNA demethylation: mechanism, function and beyond”. Nature Reviews Genetics 18 (9): 517–534. doi :10.1038/nrg.2017.33 . ISSN 1471-0056 . PMID 28555658 . ^ Song, Chun-Xiao; Szulwach, Keith E.; Dai, Qing; Fu, Ye; Mao, Shi-Qing; Lin, Li; Street, Craig; Li, Yujing et al. (2013-04-25). “Genome-wide profiling of 5-formylcytosine reveals its roles in epigenetic priming” . Cell 153 (3): 678–691. doi :10.1016/j.cell.2013.04.001 . ISSN 1097-4172 . PMC 3657391 . PMID 23602153 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC3657391/ . ^ Ito, Shinsuke; Shen, Li; Dai, Qing; Wu, Susan C.; Collins, Leonard B.; Swenberg, James A.; He, Chuan; Zhang, Yi (2011-09-02). “Tet Proteins Can Convert 5-Methylcytosine to 5-Formylcytosine and 5-Carboxylcytosine” (英語). Science 333 (6047): 1300–1303. Bibcode : 2011Sci...333.1300I . doi :10.1126/science.1210597 . ISSN 0036-8075 . PMC 3495246 . PMID 21778364 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC3495246/ . ^ Lu, Xingyu; Zhao, Boxuan Simen; He, Chuan (2015-02-12). “TET Family Proteins: Oxidation Activity, Interacting Molecules, and Functions in Diseases” . Chemical Reviews 115 (6): 2225–2239. doi :10.1021/cr500470n . ISSN 0009-2665 . PMC 4784441 . PMID 25675246 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4784441/ . ^ Xu, Wei; Yang, Hui; Liu, Ying; Yang, Ying; Wang, Ping; Kim, Se-Hee; Ito, Shinsuke; Yang, Chen et al. (2011-01-18). “Oncometabolite 2-Hydroxyglutarate Is a Competitive Inhibitor of α-Ketoglutarate-Dependent Dioxygenases” . Cancer Cell 19 (1): 17–30. doi :10.1016/j.ccr.2010.12.014 . ISSN 1535-6108 . PMC 3229304 . PMID 21251613 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC3229304/ . ^ a b c Ehrlich, Melanie (2009-12-01). “DNA hypomethylation in cancer cells” . Epigenomics 1 (2): 239–259. doi :10.2217/epi.09.33 . ISSN 1750-1911 . PMC 2873040 . PMID 20495664 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC2873040/ .
^ Jones, Peter A. (1996-06-01). “DNA Methylation Errors and Cancer” (英語). Cancer Research 56 (11): 2463–2467. ISSN 0008-5472 . PMID 8653676 . https://cancerres.aacrjournals.org/content/56/11/2463 . ^ Robertson, K D; Uzvolgyi, E; Liang, G; Talmadge, C; Sumegi, J; Gonzales, F A; Jones, P A (1999-06-01). “The human DNA methyltransferases (DNMTs) 1, 3a and 3b: coordinate mRNA expression in normal tissues and overexpression in tumors.” . Nucleic Acids Research 27 (11): 2291–2298. doi :10.1093/nar/27.11.2291 . ISSN 0305-1048 . PMC 148793 . PMID 10325416 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC148793/ . ^ Hanahan, Douglas; Weinberg, Robert A. (2011-03-04). “Hallmarks of Cancer: The Next Generation” (English). Cell 144 (5): 646–674. doi :10.1016/j.cell.2011.02.013 . ISSN 0092-8674 . PMID 21376230 . https://www.cell.com/cell/abstract/S0092-8674(11)00127-9 . ^ Horvath, Steve; Raj, Kenneth (June 2018). “DNA methylation-based biomarkers and the epigenetic clock theory of ageing” (英語). Nature Reviews Genetics 19 (6): 371–384. doi :10.1038/s41576-018-0004-3 . ISSN 1471-0064 . PMID 29643443 . ^ Horvath, Steve (2013-12-10). “DNA methylation age of human tissues and cell types” . Genome Biology 14 (10): 3156. doi :10.1186/gb-2013-14-10-r115 . ISSN 1474-760X . PMC 4015143 . PMID 24138928 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4015143/ . ^ Hannum, Gregory; Guinney, Justin; Zhao, Ling; Zhang, Li; Hughes, Guy; Sadda, SriniVas; Klotzle, Brandy; Bibikova, Marina et al. (2013-01-24). “Genome-wide Methylation Profiles Reveal Quantitative Views of Human Aging Rates” . Molecular Cell 49 (2): 359–367. doi :10.1016/j.molcel.2012.10.016 . ISSN 1097-2765 . PMC 3780611 . PMID 23177740 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC3780611/ . ^ Levine, Morgan E.; Lu, Ake T.; Quach, Austin; Chen, Brian H.; Assimes, Themistocles L.; Bandinelli, Stefania; Hou, Lifang; Baccarelli, Andrea A. et al. (2018-04-17). “An epigenetic biomarker of aging for lifespan and healthspan” . Aging (Albany NY) 10 (4): 573–591. doi :10.18632/aging.101414 . ISSN 1945-4589 . PMC 5940111 . PMID 29676998 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC5940111/ . ^ Garagnani, Paolo; Bacalini, Maria G.; Pirazzini, Chiara; Gori, Davide; Giuliani, Cristina; Mari, Daniela; Blasio, Anna M. Di; Gentilini, Davide et al. (2012). “Methylation of ELOVL2 gene as a new epigenetic marker of age” (英語). Aging Cell 11 (6): 1132–1134. doi :10.1111/acel.12005 . ISSN 1474-9726 . PMID 23061750 .
Information related to 5-メチルシトシン