|
Vírus Ebola
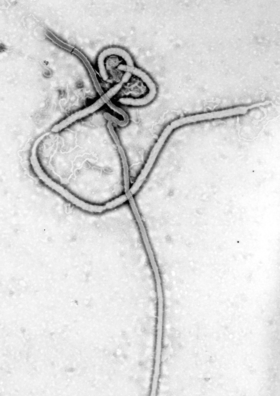
Vírus Ebola (português brasileiro) ou Vírus Ébola (português europeu)[1] (EBOV, anteriormente designado Ebolavírus Zaire) é o único membro da espécie Zaire ebolavírus e o mais perigoso dos cinco vírus conhecidos dentro do gênero Ebolavírus.[2] Quatro dos cinco ebolavírus conhecidos causam febre e uma hemorragia grave e muitas vezes fatal em seres humanos e outros mamíferos, patologia conhecida febre hemorrágica Ebola. O vírus e suas espécies foram originalmente chamados de Zaire (hoje República Democrática do Congo), o país onde foi descrito pela primeira vez[2] e quando houve a primeira suspeita de ser uma nova "estirpe" do vírus de Marburg, intimamente relacionado;[3][4][4] o vírus (mas não a sua espécie) foi renomeado para "vírus Ebola", em 2010, para evitar confusão. A espécie é uma espécie de táxon virológico incluído no gênero Ebolavirus, família Filoviridae (cujos membros são chamados de Filovírus), ordem Mononegavirales.[2] Acredita-se que seu reservatório natural sejam morcegos, particularmente morcegos frugívoros, e é transmitida principalmente entre os seres humanos e de animais para humanos, através de fluidos corporais. O vírus Ebola causou a maioria das mortes humanas por doença por vírus Ébola (DVE) e foi a causa da epidemia de 2013-2016 na África Ocidental,[5] que resultou em pelo menos 28.646 casos suspeitos e 11.323 mortes confirmadas.[6] O genoma do EBOV é de aproximadamente 19.000 nucleotídeos. Ele codifica sete proteínas estruturais:. nucleoproteína (NP), polimerase cofator (VP35), (VP40), GP, ativador de transcrição (VP30), VP24 e ARN-polimerase (L).[7] Devido à sua alta taxa de mortalidade, o EBOV também é listado como um agente de seleção. Foi classificado como um patógeno de Risco 4 pela Organização Mundial de Saúde (o que exige Nível de Biossegurança 4 contenção equivalente); um patógeno de Categoria A pelos Institutos Nacionais da Saúde/Instituto Nacional de Alergia e Doenças Infecciosas; um agente de bioterrorismo de Categoria A pelo Centro de Controle e Prevenção de Doenças; além de ser listado pelo Grupo Austrália como um agente biológico que deve ser controlado. Estrutura  O EBOV carrega um genoma de RNA de sentido negativo em vírions que são cilíndricos/tubulares e contêm componentes de envelope viral, matriz e nucleocapsídeo. O vírus geralmente possui aproximadamente 80 nm de diâmetro e 970 nm de comprimento e possui uma glicoproteína (GP) codificada pelo vírus que se projeta como espículas de 7 a 10 nm de comprimento a partir de sua superfície de bicamada lipídica.[8] O envelope viral do vírion é derivado por brotamento dos domínios da membrana da célula hospedeira nos quais as espículas de GP foram inseridas durante sua biossíntese. As moléculas individuais de GP aparecem com espaçamentos de cerca de 10 nm. As proteínas virais VP40 e VP24 estão localizadas entre o envelope e o nucleocapsídeo (veja a seguir), no espaço da matriz.[9] No centro da estrutura do vírion está o nucleocapsídeo, que é composto por uma série de proteínas virais ligadas a um RNA de sentido negativo linear de 18–19 kb, sem poliadenilação, de 3′ ou 5′ (veja a seguir); o RNA é enrolado helicoidalmente e complexado com as proteínas NP, VP35, VP30 e L. Essa hélice tem um diâmetro de 80 nm.[10][10][11] A forma geral dos vírions após a purificação e a visualização (por exemplo, por ultracentrifugação e microscopia eletrônica, respectivamente) varia consideravelmente; cilindros simples são muito menos prevalentes do que estruturas em direção inversa, ramificações e loops (por exemplo, formas de cajado U, de parafuso olhal 9 ou outras aparências circulares/enroladas), cuja origem pode estar nas técnicas laboratoriais aplicadas.[12][13] A estrutura "filiforme" característica é, no entanto, uma característica morfológica mais geral dos filovírus (juntamente com seu envelope viral com glicoproteína, nucleocapsídeo de RNA, etc.).[12] GenomaCada vírion contém uma molécula de RNA linear, de fita simples e sentido negativo, com 18.959 a 18.961 nucleotídeos de comprimento.[14] A extremidade 3′ não é poliadenilada e a extremidade 5′ não possui cap. Esse genoma viral codifica sete proteínas estruturais e uma proteína não estrutural. A ordem dos genes é 3′ - leader - NP - VP35 - VP40 - GP/sGP - VP30 - VP24 - L - trailer - 5′; sendo que o leader e o trailer são regiões não transcritas, que carregam sinais importantes para controlar a transcrição, a replicação e o empacotamento dos genomas virais em novos vírions. As seções dos genes NP, VP35 e L dos filovírus foram identificadas como endógenas nos genomas de vários grupos de pequenos mamíferos.[15][16][17]  Descobriu-se que 472 nucleotídeos da extremidade 3' e 731 nucleotídeos da extremidade 5' são suficientes para a replicação de um "minigenoma" viral, embora não sejam suficientes para a infecção.[12] O sequenciamento do vírus de 78 pacientes com doença confirmada pelo vírus Ebola, representando mais de 70% dos casos diagnosticados em Serra Leoa do final de maio a meados de junho de 2014,[18][19] forneceu evidências de que o surto de 2014 não estava mais sendo alimentado por novos contatos com seu seu reservatório natural. Usando a tecnologia de sequenciamento de terceira geração, os pesquisadores conseguiram sequenciar amostras em apenas 48 horas.[20] Como outros vírus de RNA,[18] o vírus Ebola sofre mutação rapidamente, tanto dentro de uma pessoa durante a progressão da doença quanto no reservatório entre a população humana local.[19] A taxa de mutação observada de 2,0 x 10−3 substituições por local por ano é tão rápida quanto a da gripe sazonal.[21]
Entrada Há dois candidatos a proteínas de entrada na célula hospedeira. A primeira é uma proteína transportadora de colesterol, a C1 de Niemann-Pick (NPC1) codificada pelo hospedeiro, que parece ser essencial para a entrada dos vírions do Ebola na célula hospedeira e para sua replicação final.[22][23] Em um estudo, camundongos com uma cópia do gene NPC1 inativado apresentaram uma taxa de sobrevivência de 80% quinze dias após a exposição ao vírus Ebola adaptado para camundongos, enquanto apenas 10% dos camundongos não modificados sobreviveram por esse tempo.[22] Em outro estudo, foi demonstrado que micromoléculas inibem a infecção pelo vírus Ebola ao impedir que a glicoproteína (GP) do envelope viral se ligue à NPC1.[23][24] Portanto, a NPC1 demonstrou ser essencial para a entrada desse filovírus, pois medeia a infecção ligando-se diretamente à GP viral.[23] Quando as células de indivíduos com Doença de Niemann-Pick (que não tinham o transportador NPC1) foram expostas ao vírus Ebola em laboratório, as células sobreviveram e pareciam impermeáveis ao vírus, indicando ainda que o Ebola depende do NPC1 para entrar nas células.[22] Mutações no gene NPC1 em humanos foram conjecturadas como um possível modo de tornar alguns indivíduos resistentes a essa doença viral mortal. Os mesmos estudos descreveram resultados semelhantes em relação à função do NPC1 para o vírus de Marburg, um filovírus semelhante.[22] Um outro estudo também apresentou evidências de que o gene NPC1 é o receptor crítico que medeia a infecção pelo Ebola por meio de sua ligação direta à GP viral e que é o segundo domínio "lisossômico" do NPC1 que medeia essa ligação.[25] O segundo candidato é o TIM-1 (também conhecido como HAVCR1).[26] Foi demonstrado que o TIM-1 se liga ao domínio de ligação do receptor da glicoproteína do EBOV para aumentar a receptividade das células Vero. O bloqueio do seu efeito com siRNA impediu a infecção das células Vero. O TIM1 é expresso em tecidos conhecidos por serem seriamente afetados pela lise do EBOV (traqueia, córnea e conjuntiva). Um anticorpo monoclonal contra o domínio IgV do TIM-1, ARD5, bloqueou a ligação e a infecção pelo EBOV. Juntos, esses estudos sugerem que o NPC1 e o TIM-1 podem ser alvos terapêuticos em potencial para um medicamento antiviral contra o Ebola e como base para um ensaio de diagnóstico de campo rápido. Replicação Por serem acelulares, os vírus como o Ebola não se replicam por meio de nenhum tipo de divisão celular; em vez disso, eles usam uma combinação de enzimas codificadas pelo hospedeiro e pelo vírus, juntamente com estruturas da célula hospedeira, para produzir várias cópias de si mesmos. Em seguida, essas cópias se automontam em estruturas macromoleculares virais na célula hospedeira.[27] O vírus conclui um conjunto de etapas ao infectar cada célula individual. O vírus começa seu ataque ligando-se aos receptores do hospedeiro por meio do peplômero da superfície da glicoproteína (GP) e é endocitado em macropinossomos na célula hospedeira.[28] Para penetrar na célula, a membrana viral se funde com a membrana da vesícula e o nucleocapsídeo é liberado no citoplasma. O ssRNA genômico de sentido negativo encapsidado é usado como modelo para a síntese (3'-5') de mRNAs monocistrônicos poliadenilados e, usando os ribossomos da célula hospedeira, moléculas de tRNA, etc., o mRNA é traduzido em proteínas virais individuais.[29][30][31] Essas proteínas virais são processadas: um precursor de glicoproteína (GP0) é clivado em GP1 e GP2, que são então fortemente glicosiladas usando enzimas e substratos celulares. Essas duas moléculas se reúnem, primeiro em heterodímeros e, depois, em trímeros para formar os peplômeros de superfície. O precursor da glicoproteína segregada secretada (sGP) é clivado em sGP e polipeptídeo (polipeptídeo), ambos liberados da célula. À medida que os níveis de proteína viral aumentam, ocorre uma mudança da tradução para a replicação. Usando o RNA genômico de sentido negativo como modelo, um +ssRNA complementar é sintetizado; esse é então usado como modelo para a síntese do novo (-)ssRNA genômico, que é rapidamente encapsidado. Os nucleocapsídeos recém-formados e as proteínas do envelope se associam à membrana plasmática da célula hospedeira e ocorre a excreção viral, destruindo a célula. EcologiaO vírus Ebola é um patógeno zoonótico. Foi relatado que os hospedeiros intermediários são "várias espécies de morcegos frugívoros...em toda a África central e subsariana". A evidência de infecção em morcegos foi detectada por meios moleculares e sorológicos. No entanto, o vírus não foi isolado em morcegos.[32][33] Os hospedeiros finais são humanos e grandes primatas, infectados por contato com morcegos ou por outros hospedeiros finais. Foi relatado que porcos nas Filipinas foram infectados com o vírus Reston, portanto, podem existir outros hospedeiros intermediários ou amplificadores.[33] Os surtos do vírus Ebola tendem a ocorrer quando as temperaturas são mais baixas e a umidade é mais alta do que o normal na África.[34] Mesmo depois que uma pessoa se recupera da fase aguda da doença, o vírus Ebola sobrevive por meses em determinados órgãos, como olhos e testículos.[35] Doença por vírus EbolaO Zaire ebolavírus é um dos quatro vírus Ebola conhecidos por causar doenças em humanos. Ele tem a maior taxa de letalidade entre esses vírus, com uma média de 83% desde os primeiros surtos em 1976, embora uma taxa de letalidade de até 90% tenha sido registrada em um surto na República do Congo entre dezembro de 2002 e abril de 2003. Também houve mais surtos do Zaire ebolavírus do que de qualquer outro vírus Ebola. O primeiro surto ocorreu em 26 de agosto de 1976 em Yambuku.[36] O primeiro caso registrado foi o de Mabalo Lokela, um professor de 44 anos. Os sintomas se assemelhavam aos da malária, e os pacientes subsequentes receberam quinino. A transmissão foi atribuída à reutilização de agulhas não esterilizadas e ao contato pessoal próximo, fluidos corporais e locais onde a pessoa tocou. Durante o surto de Ebola de 1976 no Zaire, Ngoy Mushola viajou de Bumba para Yambuku, onde registrou a primeira descrição clínica da doença em seu diário:
Desde a primeira descrição clínica registrada da doença em 1976 no Zaire, o surto de Ebola, que começou em março de 2014, atingiu proporções epidêmicas e matou mais de 8.000 pessoas em janeiro de 2015. Esse surto foi centrado na África Ocidental, uma área que não havia sido afetada anteriormente pela doença. O número de vítimas foi particularmente grave em três países: Guiné, Libéria e Serra Leoa. Alguns casos também foram relatados em países fora da África Ocidental, todos relacionados a viajantes internacionais que foram expostos nas regiões mais afetadas e posteriormente apresentaram sintomas da febre Ebola após chegarem a seus destinos.[38] A gravidade da doença em humanos varia amplamente, desde a rápida fatalidade até a doença leve ou até mesmo a resposta assintomática.[39] Estudos de surtos no final do século XX não conseguiram encontrar uma correlação entre a gravidade da doença e a natureza genética do vírus. Portanto, suspeitou-se que a variabilidade na gravidade da doença estivesse correlacionada com as diferenças genéticas das vítimas. Tem sido difícil estudar isso em modelos animais que respondem ao vírus com febre hemorrágica de maneira semelhante à dos seres humanos, porque os modelos típicos de camundongos não respondem dessa forma e o grande número necessário de cobaias apropriadas não está facilmente disponível. No final de outubro de 2014, uma publicação relatou um estudo da resposta a uma cepa adaptada de camundongo do Zaire ebolavírus apresentada por uma população geneticamente diversa de camundongos que foi criada para ter uma gama de respostas ao vírus que inclui a fatalidade da febre hemorrágica.[40] VacinaEm dezembro de 2016, um estudo constatou que a vacina VSV-EBOV era 70-100% eficaz contra o vírus do Zaire (não o do Sudão), tornando-a a primeira vacina contra a doença.[41][42] A VSV-EBOV foi aprovada pela Administração de Alimentos e Medicamentos dos EUA (FDA) em dezembro de 2019.[43] História e nomenclatura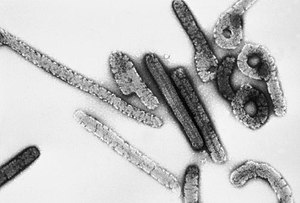 O vírus Ebola foi identificado pela primeira vez como uma possível nova "cepa" do vírus de Marburg em 1976.[3][4][44] O Comitê Internacional de Taxonomia de Vírus (ICTV) identifica o vírus Ebola como a espécie Zaire ebolavirus, que faz parte do gênero Ebolavirus, família Filoviridae, ordem Mononegavirales. O nome "vírus Ebola" é derivado do rio Ebola - um rio que, a princípio, acreditava-se estar próximo à área da República Democrática do Congo, anteriormente chamada de Zaire, onde ocorreu o surto do vírus Ebola do Zaire em 1976 - e do sufixo taxonômico virus.[2][3][4][45] Em 1998, o nome do vírus foi alterado para "Zaire Ebola virus"[46][47] e, em 2002, para a espécie Zaire ebolavirus.[48][49] Entretanto, a maioria dos artigos científicos continuou a se referir ao "vírus Ebola" ou usou os termos "vírus Ebola" e "Zaire ebolavirus" em paralelo. Consequentemente, em 2010, um grupo de pesquisadores recomendou que o nome "vírus Ebola" fosse adotado para uma subclassificação dentro da espécie Zaire ebolavirus, com a abreviação EBOV correspondente.[2] As abreviações anteriores para o vírus eram EBOV-Z (para "vírus Ebola Zaire") e ZEBOV (para "vírus Ebola Zaire" ou "Zaire ebolavirus"). Em 2011, o ICTV rejeitou explicitamente uma proposta (2010.010bV) para reconhecer esse nome, pois o ICTV não designa nomes para subtipos, variantes, cepas ou outros agrupamentos em nível de subespécie.[50] Atualmente, o ICTV não reconhece oficialmente o "vírus Ebola" como uma classificação taxonômica, mas continua a usar e recomendar apenas a designação de espécie Zaire ebolavirus.[51] O tipo nomenclatural do vírus Ebola, variante Mayinga (EBOV/May), foi nomeado em homenagem a Mayinga N'Seka, uma enfermeira que morreu durante o surto de 1976 no Zaire.[2][52][53] O nome Zaire ebolavirus é derivado de Zaire e do sufixo taxonômico ebolavirus (que denota uma espécie de ebolavírus e se refere ao rio Ebola).[2] De acordo com as regras de nomenclatura de táxons estabelecidas pelo Comitê Internacional de Taxonomia de Vírus (ICTV), o nome Zaire ebolavirus deve ser sempre maiúsculo, em itálico e precedido pela palavra "espécie". Os nomes de seus membros (Zaire ebolavirus) devem ser capitalizados, não devem estar em itálico e devem ser usados sem artigos.[2] Critérios de inclusão de vírusUm vírus do gênero Ebolavirus é um membro da espécie Zaire ebolavirus se:[2]
EvoluçãoA diversidade genética do Zaire ebolavirus divergiu de seus ancestrais entre 1960 e 1976.[54] A diversidade genética do ebolavírus permaneceu constante antes de 1900.[54][55] Então, por volta da década de 1960, provavelmente devido à mudança climática ou às atividades humanas, a diversidade genética do vírus caiu rapidamente e a maioria das linhagens foi extinta.[55] À medida que o número de hospedeiros suscetíveis diminui, o mesmo acontece com o tamanho efetivo da população e sua diversidade genética. Esse efeito de funil genético tem implicações para a capacidade da espécie de causar a doença por vírus Ebola em hospedeiros humanos. Um evento de recombinação entre as linhagens do Zaire ebolavirus provavelmente ocorreu entre 1996 e 2001 em macacos selvagens, dando origem a vírus descendentes recombinantes.[56] Esses vírus recombinantes foram supostamente responsáveis por uma série de surtos entre humanos na África Central em 2001-2003.[56] O Zaire ebolavirus - variante Makona causou o surto de 2014 na África Ocidental.[57] O surto foi caracterizado pela mais longa instância de transmissão de humano para humano da espécie viral.[57] Pressões para se adaptar ao hospedeiro humano foram observadas nesse momento, no entanto, nenhuma mudança fenotípica no vírus (como aumento da transmissão, aumento da evasão imunológica pelo vírus) foi observada. Ver tambémReferências
Bibliografia
Ligações externas
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||